中国病毒学论坛|我们一直在坚持!
标题: 华人学者惊人发现:HIV可以逃避CRISPR/Cas9治疗 [打印本页]
作者: ipsvirus 时间: 2016-4-10 16:25
标题: 华人学者惊人发现:HIV可以逃避CRISPR/Cas9治疗
本帖最后由 ipsvirus 于 2016-4-10 16:26 编辑
《Cell Reports》上的一项研究,在将CRISPR/Cas9用作有效的抗病毒药物之前,或许还需要对这一基因编辑平台进行稍微的调整。利用CRISPR/Cas9突变细胞DNA内HIV-1的研究人员发现,尽管单突变可以抑制病毒复制,一些也可以导致意外的抵抗。
当前CRISPR/Cas9已被成功改造成基因组定点编辑工具。因此,如何利用CRISPR/Cas9系统对HIV-1病毒基因进行高效靶向修饰,从而达到治疗HIV-1感染病患的目的成为了国内外许多实验室的研究热点。
2015年10月,来自吉林大学和斯坦福大学的研究人员,利用一种工程设计的CRISPR Csy4 RNA内切核糖核酸酶,来抑制HIV-1病毒感染。相关研究结果发表在国际学术期刊《PLOS ONE》上。
哺乳动物细胞编码的限制因子,可以限制HIV-1和其他病毒的复制。不过,被病毒感染的细胞往往不表达这样的限制因子。人们普遍认为,诱导宿主限制因子的表达是抑制病毒复制的一个潜在途径。杜克大学的Bryan R. Culle领导研究团队在CRISPR/Cas9的基础上构建了转录激活子,成功让人类细胞表达了自己缺乏的限制因子(A3G和A3B)。这项研究发表在2015年12月的PNAS杂志上。
HIV是一种有效和狡猾的逆转录病毒。一旦HIV将其DNA导入宿主细胞基因组,它就有一段很长的潜伏期,并能保持休眠状态潜伏很多年。虽然医生可以调制相匹配的抗逆转录病毒药物鸡尾酒,来抑制病毒,但是如果停止治疗病毒又会再度复活。为了使潜伏性HIV变得完全无害,美国麻省大学医学院的研究人员利用Cas9 / CRISPR——一种强大的基因编辑工具,开发出一种新型技术,有可能将潜在病毒的DNA从被感染细胞中去除。
然而,根据发表在4月7日《Cell Reports》杂志上的一项研究,在将CRISPR/Cas9用作有效的抗病毒药物之前,或许还需要对这一基因编辑平台进行稍微的调整。利用CRISPR/Cas9突变细胞DNA内HIV-1的研究人员发现,尽管单突变可以抑制病毒复制,一些也可以导致意外的抵抗。研究人员认为,靶向多个病毒DNA区域对于有效实现CRISPR/Cas9的潜在抗病毒能力或许是必要的。
一旦进入到细胞中,HIV的RNA基因组会转变为DNA,与细胞DNA缠绕在一起。由此可以编程CRISPR/Cas9来靶向一段DNA序列,切割病毒DNA。问题是,HIV极擅长携带新突变存活及旺盛生长,因此尽管许多的病毒被这种靶向方法所杀死,逃避CRISPR/Cas9治疗的HIV会变得更难靶向。
论文的资深作者、麦吉尔大学艾滋病中心副教授、犹太综合医院Lady Davis研究所的高级研究员梁晨(Chen Liang,音译)说:“当我们测序逃脱HIV的病毒RNA时,惊讶地发现病毒的大多数突变都恰好排列在Cas9切割DNA的位点,这直接表明了这些突变是修复断裂DNA时细胞非同源末端连接机器所介导,而非病毒逆转录酶的错误所致。”
“一些突变很小,只有单核苷酸,但这一突变改变了序列,使得Cas9不再能够识别它。这样的突变不会损伤病毒,因此这些抵抗病毒仍然可以复制。”
这项由麦吉尔大学、蒙特利尔大学、中国医学科学院和北京协和医学院的研究人员协作完成的研究,为希望将CRISPR/Cas9用作为一种抗病毒药物的人提供了一个警示故事。梁晨不认为这些努力是徒劳的,因为有一些策略可以克服这一限制。例如,利用CRISPR/Cas9或Cas9以外的其他酶来靶向多个位点。一旦确定了一种解决方案,下一个障碍将是找到一些方法向患者递送治疗物。
梁晨说:“不仅是HIV-1,CRISPR/Cas9为许多其他的病毒找到一种治愈方法带来了新希望。朝着这一目标,我们还有一条漫长的路要走,有许多的障碍和限制需要我们去克服,但我们有信息将会获得成功。”
来源:生物探索
作者: ipsvirus 时间: 2016-4-10 16:26
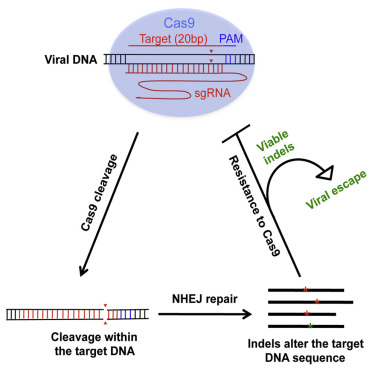
CRISPR/Cas9-Derived Mutations Both Inhibit HIV-1 Replication and Accelerate Viral Escape
Zhen Wang, Qinghua Pan, Patrick Gendron, Weijun Zhu, Fei Guo, Shan Cen, Mark A. Wainberg, Chen Liang
Highlights
•Cas9/sgRNA suppresses HIV-1 replication
•HIV-1 escapes from inhibition mediated by Cas9/sgRNA
•Escape mutations are located to the Cas9 cleavage site within the target viral DNA
•Cas9/sgRNA-induced mutations assist viral escape
Summary
Cas9 cleaves specific DNA sequences with the assistance of a programmable single guide RNA (sgRNA). Repairing this broken DNA by the cell’s error-prone non-homologous end joining (NHEJ) machinery leads to insertions and deletions (indels) that often impair DNA function. Using HIV-1, we have now demonstrated that many of these indels are indeed lethal for the virus, but that others lead to the emergence of replication competent viruses that are resistant to Cas9/sgRNA. This unexpected contribution of Cas9 to the development of viral resistance is facilitated by some indels that are not deleterious for viral replication, but that are refractory to recognition by the same sgRNA as a result of changing the target DNA sequences. This observation illustrates two opposite outcomes of Cas9/sgRNA action, i.e., inactivation of HIV-1 and acceleration of viral escape, thereby potentially limiting the use of Cas9/sgRNA in HIV-1 therapy.
http://www.cell.com/cell-reports/abstract/S2211-1247(16)30298-4
作者: 序道 时间: 2016-4-11 08:01
不错

| 欢迎光临 中国病毒学论坛|我们一直在坚持! (http://bbs.virology.com.cn/) |
Powered by Discuz! X3.2 |